Polymerase Chain Reaction (Amplifikasi DNA)
PCR Primer
Meskipun PCR merupakan teknik yang luar biasa, proses PCR dapat gagal jika primer yang dirancang salah. Primer PCR harus mampu mengikat DNA target spesifik dengan cukup lama, dan harus secara spesifik mengikat DNA target sebagai bagian dari komponen tersebut. Program komputer telah dikembangkan untuk membantu mendesain DNA primer PCR dan telah tersedia pada berbagai sumber di internet. Spesifisitas primer PCR adalah salah satu karakteristik yang paling penting dan sebagian besar disebabkan oleh urutan nukleotida primer PCR. Untuk memastikan pengikatan spesifik primer hanya dengan DNA target, primer PCR harus memiliki panjang yang cukup. Primer PCR spesifik panjangnya antara 18 dan 22 nukleotida. Probabilitas primer berikatan dengan urutan acak DNA template akan menurun seiring dengan bertambahnya panjang primer.
Latihan sederhana dalam mengukur probabilitas primer menempel pada DNA template dapat menggunakan konsep berikut ini. Pertama, asumsikan bahwa sampel DNA genom memiliki jumlah G, A, T, dan C yang sama, maka probabilitas nukleotida spesifik untuk masing-masing nukleotida adalah 0,25. Dengan kata lain, pada posisi apa pun dalam genom, ada satu dari empat kemungkinan bahwa itu diposisi tersebut terdapat basa nitrogen Adenin. Demikian pula, posisi apa pun dalam DNA akan memiliki satu dari empat peluang menjadi C, T, atau G. Urutan seperti itu terjadi karena GATC memiliki probabilitas (0,25) x (0,25) x (0,25) x (0,25) = 0,0039. Dalam genom manusia ada 3,3 miliar nukleotida, sehingga urutan GATC kemungkinan akan muncul 12.870.000 kali. Sebaliknya, panjang nukleotida urutan 20 memiliki peluang (0,25)20 = 9,09 x 1013. Kemungkinan bahwa 20 urutan nukleotida akan terjadi secara acak dalam genom sebenarnya 0,003, kurang dari 1. Oleh karena itu, primer dari 20 Nukleotida yang dirancang untuk melengkapi DNA target dalam genom memiliki kemungkinan sangat rendah untuk mengikat ke urutan lain. Tentu saja latihan ini memiliki beberapa asumsi. Pertama, genom manusia memiliki banyak sekuens DNA yang berulang dan merancang primer untuk mengikat pengulangan berarti juga dapat mengikat sekuen berulang lainnya, bahkan jika panjangnya 20 nukleotida. Selain itu, probabilitas mengasumsikan bahwa genom akan memiliki G, A, T, dan C dengan jumlah yang sama. Pada kenyataannya, sebagian besar genom memiliki lebih banyak pasangan basa A & T daripada pasangan basa G & C, atau sebaliknya.
Selain panjang, urutan nukleotida DNA primer merupakan karakteristik desain yang penting dari sebuah primer. Urutan harus saling melengkapi dan sesuai dengan target DNA yang akan diamplifikasi. Jumlah keseluruhan G, A, T, dan C dalam primer dan urutan spesifik dari basa ini juga mempengaruhi efisiensi PCR secara keseluruhan.
Urutan nukleotida primer PCR merupakan penentu utama suhu leleh primer atau TM (Temperature Melting). TM untuk primer adalah suhu di mana setengah dari primer berikatan hidrogen dengan urutan komplementer dari DNA target dan setengah lainnya tidak terikat. TM umumnya berbeda dari satu primer ke primer lainnya, karena pasangan basa A & T disatukan oleh dua ikatan hidrogen, sedangkan pasangan basa G & C disatukan dengan tiga ikatan hidrogen. Semakin banyak G atau C dalam primer, semakin banyak pula ikatan hidrogen, dan oleh karena itu, lebih tinggi suhu yang diperlukan untuk memisahkan primer dari DNA targetnya. Tetapi parameter ini adalah salah satu faktor untuk menghitung TM. Selain itu, konsentrasi garam di lingkungan primer dan DNA target, konsentrasi primer dan DNA target, serta interaksi antara basa di dalam primer itu sendiri, akan mempengaruhi TM DNA primer. Perhitungan TM biasanya ditentukan oleh algoritma komputer, banyak di antaranya tersedia secara online. Alasan bahwa TM harus dihitung secara akurat adalah karena suhu annealing untuk reaksi biasanya pada suhu 5 derajat Celcius di bawah TM terendah untuk primer forward ataupun reverse. Jika suhu annealing ditetapkan terlalu tinggi, maka primer mungkin tidak menempel ke DNA target, sehingga tidak akan mengalami elongasi atau extension. Di sisi lain, jika suhu annealing ditetapkan terlalu rendah, primer PCR dapat berikatan dengan urutan yang sama di luar DNA target. Ketika terlalu rendah, primer PCR dapat menempel pada DNA dari situs yang salah. Hal Ini dapat terjadi jika hanya sebagian dari primer PCR mengikat ke daerah komplementer di luar DNA target, dan karena suhunya terlalu rendah, primer yang di annealing cukup stabil untuk enzim DNA polimerase memulai sintesis DNA dari urutan yang salah. Selain menetapkan suhu annealing dengan benar, kedua DNA primer PCR (forward & reverse) harus memiliki TM yang cukup sama, jika tidak, satu primer akan mengikat DNA target pada suhu annealing yang ditetapkan, dan yang lainnya mungkin menempel pada DNA target. Hal ini tidak akan menghasilkan amplifikasi DNA target.
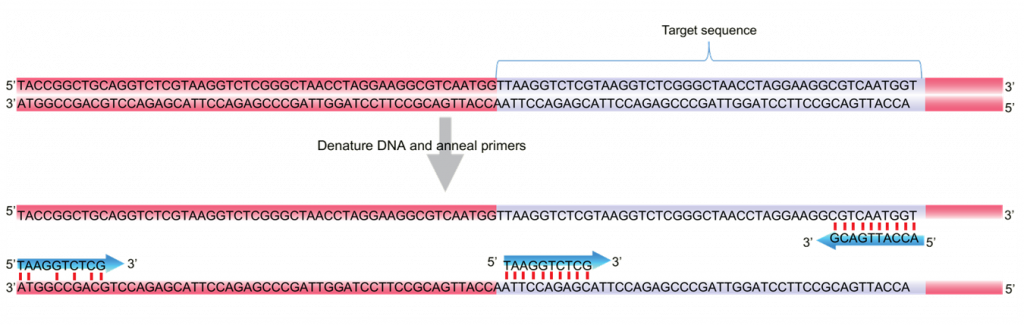
Gambar 6. Jika suhu annealing terlalu rendah, Primer PCR dapat menempel pada wilayah DNA lainnya yang bukan target. Dalam hal ini, primer forward hanya sebagian yang annealing diluar urutan target. Jika suhu annealing ditetapkan terlalu rendah, akan membuat produk PCR yang lebih panjang dari yang dirancang.
DNA Primer PCR juga harus memiliki kandungan GC antara 40% hingga 60%, yang berarti harus ada jumlah G dan C yang cukup sama dibandingkan dengan A dan T di seluruh panjang primer. Distribusi basa juga menjadi faktor penting, dan basa-basa yang berulang dapat mengacaukan fungsi primer serta menimbulkan masalah selama sintesis DNA oleh primer menggunakan enzim DNA Polymerase.
Urutan antara kedua primer juga dapat menyebabkan masalah. Jika primer forward dan reverse memiliki basa pelengkap, terutama di ujung 3’ primer, ada kemungkinan kedua primer saling menempel, yang disebut primer-dimer. Karena primer-dimer stabil, DNA polimerase dapat memulai sintesis, dan primer- dimer untai ganda yang dihasilkan diamplifikasi selama proses PCR daripada DNA target. Masalah yang diakibatkan kesalahan urutan primer PCR lainnya disebut hairpin. Hairpin terjadi ketika dua segmen DNA pada satu primer saling melengkapi, menyebabkan primer melipat ke dirinya sendiri dan bukan DNA target. Program untuk mendesain proses PCR yang tersedia secara online dapat mengidentifikasi masalah ini sehingga seorang peneliti dapat mendesain ulang primer dengan memindahkan lokasi di mana primer annealing menempel pada DNA target.
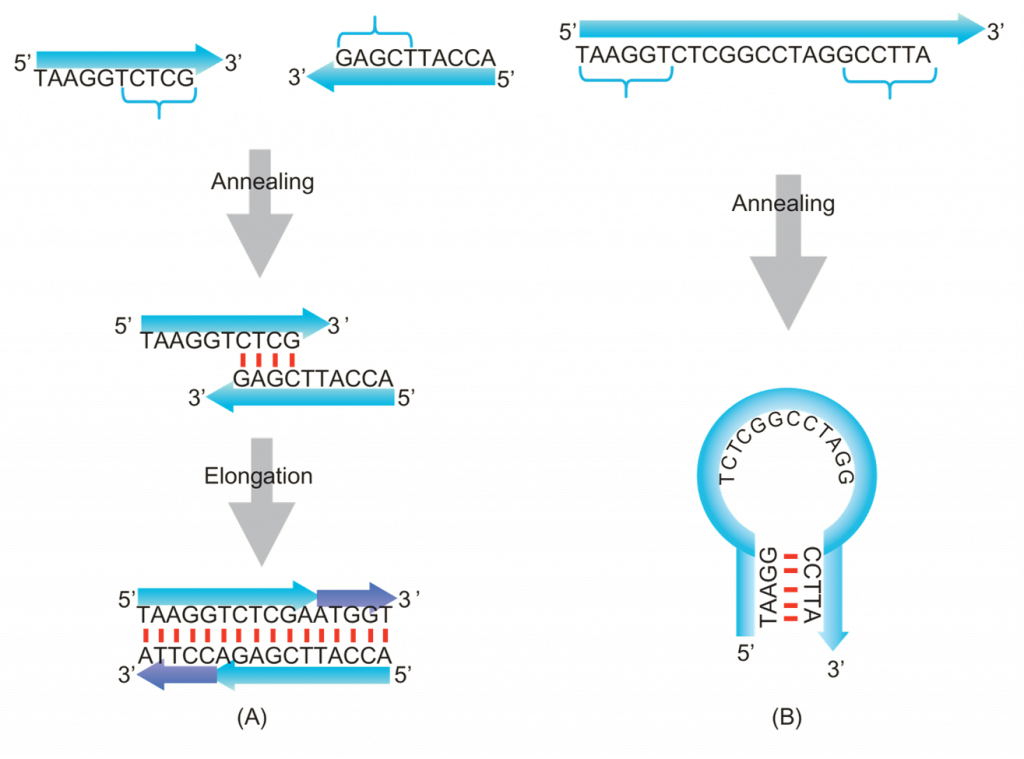
Gambar 7. Primer-dimer dan hairpin Struktur Primer PCR. Ketika urutan dalam primer PCR tunggal saling melengkapi, maka primer akan melipat kembali untuk membentuk hairpin. Jika urutan antara dua primer PCR adalah komplementer, maka primer akan mengikat antara satu sama lain dan bukan menempel pada DNA target untuk membentuk primer-dimer.
Polymerase Alternatif
Sejak diperkenalkannya Taq polimerase, berbagai enzim polimerase termostabil dari organisme termofilik lainnya juga telah digunakan dalam PCR. Pfu polimerase dari Pyrococcus furiosus digunakan ketika akan peningkatan akurasi karena memiliki kemampuan proofreading – tidak seperti Taq polimerase. TLI polimerase yang diisolasi dari Thermococcus litoralis juga memiliki 30 hingga 50 aktivitas eksonuklease proofreading untuk membuat salinan yang akurat.
PCR telah disesuaikan dengan berbagai situasi dan kondisi yang berbeda. PCR jangka panjang dapat mengamplifikasi urutan DNA dengan target yang panjang, antara 20 kb dan 30 kb. Secara normal, PCR terbatas pada panjang urutan DNA target yang berhasil diamplifikasi dan ketika urutan target lebih panjang dari 5 kb, maka tidak ada produk PCR yang terbetuk. Beberapa modifikasi sederhana digunakan untuk mengamplifikasi target panjang. Pertama, perpanjangan polimerase atau waktu extension, atau langkah ketiga dalam setiap siklus ditingkatkan. Hal ini memberi waktu bagi enzim polimerase lebih banyak waktu untuk membuat DNA. Kedua, campuran dua polimerase termostable yang berbeda. Polimerase termostabil (misalnya, Pfu atau Tli polimerase) diketahui yang memiliki kemampuan proofreading. Jika sejumlah kecil Pfu atau Tli polimerase dikombinasikan, hal ini dapat mengoreksi kesalahan apa pun yang dibuat oleh taq polymerase dan mengganti nukleotida yang salah dengan yang benar. Ketiga, urutan target yang panjang sering rusak oleh panas dan langkah denaturasi dapat menyebabkan depurination. Oleh karena itu, dalam PCR, panjang waktu denaturasi menurun dari 1 menit menjadi 2-10 detik untuk melindungi basa purin. Selain itu, suhu untuk perpanjangan atau extension lebih rendah d ari 65o C. Keempat, kondisi buffer dimodifikasi untuk memberikan konsentrasi ionik yang lebih baik untuk alternatif polimerase.


Informasi ini bagus
Assalamualaikum warahmatullahi wb kaatu Bapak Saya Rina Ariska Sihombing Nim 4203520921 PSB 20 Biologi, Saya telah membaca Materi Polymerase Chain Reaction (Amplifikasi DNA) dari yang bapak berikan,,,, saya ingin bertanya apakah Teknik PCR ini adalah teknik yang sering digunakan dalam mendiagnosis penyakit, apakah setiap untaian kerusakan pada Rantai Unit DNA dapat diatasi oleh Teknik PCR inii pak ?? Ataukah bila ada struktur sel Yang rusak dalam tubuh dapat dilihat oleh Teknik PCR pak dalam Replikasi DNA ??
Wa’alaikumsalam
Teknik PCR merupakah salah satu teknik untuk mendiagnosis penyakit, terutama untuk disease yang berkaitan dengan DNA.